研究ハイライト
植物のDNAを生きたまま観察できる手法を開発 ~ゲノム編集のハサミをランプに替えることで作物のゲノム育種に貢献~
東京理科大学理工学部応用生物科学科 松永 幸大 教授、藤本 聡 研究員、京都大学大学院理学研究科 菅野 茂夫 研究員、名古屋大学トランスフォーマティブ生命分子研究所(ITbM) 桑田 啓子 特任助教、徳島大学生物資源産業学部 刑部 敬史 教授の研究グループは、ゲノム編集技術を利用して、植物のDNAを生きたまま解析するTALE-FP法の開発に成功しました。
研究の内容:
植物のDNAを解析するためには、植物細胞を破砕して、細胞核からDNAを抽出する必要がありました。 また、植物の染色体の解析には蛍光in situハイブリダイゼーション法という手法が主に用いられていましたが、細胞を固定する必要があるため、生きた植物のDNAをリアルタイムで解析する手段はありませんでした。 近年、DNAの細胞核の中での3次元的な位置が読み取られる頻度に影響を及ぼすことが分かってきていますが、時々刻々と変わる位置情報を観察する手法が限られるため、新たな手法の開発が急務でした。
今回、本研究グループは、モデル植物であるシロイヌナズナを用いて、植物のDNAを生きた植物の組織や器官で検出するTALE-FP法を開発しました。 ゲノム編集技術で用いられているキサントモナス菌由来のTranscription activator like effector (TALE)のDNA結合ドメインに蛍光タンパク質を連結させたTALE-FPを植物体内で発現させました。 TALE-FPを検出することで、特定のDNA配列の動態を植物の様々な組織や器官で解析することに成功しました。 これにより、特定の分裂組織から染色体標本作製することなく、DNA配列の位置や分布を検出することが可能になりました。 本成果は、ゲノム育種により作成された品種選抜の高速化、植物における染色体異常の検出の簡便化など、農作物研究に大きく貢献することが期待されます。
<背景>
DNAは高度に凝縮されて細胞核に格納されています。 最近、細胞核の3次元的な位置によってDNAの情報が読み取られる頻度が変化することがわかってきました。 このため、抽出した2次元のDNAの塩基配列情報だけでなく、DNAが細胞核における3次元的な位置情報を知ることが重要となってきています。 さらに、DNAの3次元的位置は生物の発生・分化に伴い変化するほか、環境刺激や薬物によっても変化することが知られていますが、詳しいメカニズムは分かっていません。 そのため、成長や外部環境の変化に伴う植物のDNA動態メカニズムの解明が期待されていました。
ブドウ、ジャガイモ、サツマイモ、パンコムギなどの有用な作物の育種では、ゲノムを倍数化することで、果実、芋、種子の品質が優れた種の選抜が行われてきました。 このような育種の研究現場では、染色体標本を作製し、染色体の本数や形態を解析することで倍数化を確認する作業が必要です。 その解析には、特定のゲノム領域に繰り返し存在するDNA配列を用いて染色体を区別する蛍光in situハイブリダイゼーション法(FISH法)が用いられてきました。 FISH法では、植物の分裂組織から細胞壁を溶かして染色体標本を作製する必要があります。 植物の細胞壁の種類や状態に合うように、細胞壁溶解酵素の種類・活性・反応時間を調整する必要があるために、サンプル間のばらつきの大きさが問題になっていました。 また、DNA配列に蛍光標識した蛍光プローブを、熱変性した染色体標本にハイブリダイゼーション(DNA分子が相補的に結合する)することで、染色体上のDNA配列の分布を検出しますので、 ハイブリダイゼーションの条件検討に時間を要する他、検出にも2-3日の実験時間が必要でした。 そこで、染色体標本の作製やハイブリダーゼーションを行うことなく、生きた植物の組織や器官で、DNAを検出する方法の開発が期待されていました。
<内容>
本研究グループは、モデル植物であるシロイヌナズナを用いて、生きたままDNAを検出できるTALE-FP法を開発しました。 Transcription activator like effector(TALE)はキサントモナス菌由来のタンパク質で、DNA配列に特異的に結合するDNA結合タンパク質です。 このTALEにDNA分解酵素を結合することで、特定のDNA配列を切断して変異を挿入するゲノム編集は活発に研究が進んでいます。 DNA分解酵素(ハサミ)の代わりに蛍光タンパク質(ランプ)をTALEに連結させて、DNAを切断することなく、狙ったDNA配列に蛍光タンパク質を配置する方法は、動物の培養細胞において報告がありました。 しかし、動物細胞で用いられていたTALE結合型・蛍光タンパク質を用いても、植物では安定的な形質転換体を取得することが困難でした。 そこで、私達は、植物用のプロモーターを使用し、蛍光タンパク質のみを3量体にしてシグナル/ノイズ比を改善することで、安定的にDNAを検出できるTALE-FPを作成することに成功しました。
染色体の中間部分に存在するセントロメア配列、染色体の末端部分に存在するテロメア配列、タンパク質合成に関与するリボソームDNA配列を認識するTALEを設計し、緑色蛍光タンパク質(GFP)、黄色蛍光タンパク質(Venus)、赤色蛍光タンパク質(tdTomato)に連結して、植物体内で発現させました(図1)。

図1 TALE-FPの分子構造。先端に核内移行配列を持つ、中央部分のドメインはDNA配列特異的に結合する。末端に蛍光タンパク質を持つ。
その結果、花弁、めしべ、茎、葉、根などのあらゆる器官や組織の細胞核で、特定のDNA配列の蛍光シグナルを生きた細胞中に観察することができました(図2)。
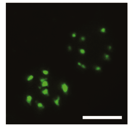
図2 セントロメアに結合したTALE-FPの蛍光写真。2つの細胞核中にTALE-FPの輝点が見える。
そして、DNA配列は細胞核内を動き回っていることを検出することができました。同じDNA配列であっても染色体の位置によって動き回る範囲が異なることもわかりました(図3)。
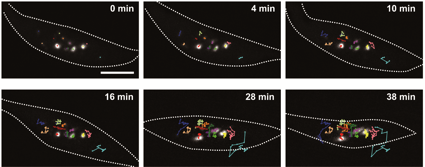
図3 細胞内を動き回るTALE-FP。白い点線は細胞核を示す。細胞核内をテロメアを認識するTALE-FPが動き回っていることがわかる。TALE-FPのそれぞれの輝点をトラッキングしたラインを示している。
このことは、植物のDNAは空間的な位置が固定されているのではなく、空間的な位置が移動しながら、遺伝子発現のスイッチのON/OFFを繰り返していることを示しています。 今後、このTALE-FPを用いて植物DNAの空間的配置を制御する分子メカニズムを解明していく予定です。
<本研究の社会的貢献>
本研究成果により、植物のDNAをあらゆる組織で生きたまま解析することができるようになりました。染色体標本を作製することなく、DNAの分布を解析できることから、ゲノム育種の研究現場における染色体解析や倍数性解析の効率化を図ることが可能になります。本法に用いたTALEは無毒化されており、どの植物にも導入可能なタンパク質であるため、今後、有用作物のDNA解析にも応用可能であると考えられます。また、植物の細胞核内におけるDNAの3次元的配置の解析や動態研究が飛躍的に進むことが期待されます。本成果により、従来法で解析することが困難であった、発生分化や環境刺激によるDNAの3次元的位置の変化のタイミングやスピードが明らかになります。最近、DNAの3次元的配置にはエピゲノム制御が関与することが示唆されています。従って、DNAの配列を操作することなくDNAの細胞核における位置を変えることで、遺伝子発現のスイッチのON/OFFを可能とするエピゲノム制御による育種にも貢献することが期待されます。
論文情報:
This article "Visualization of specific repetitive genomic sequences with fluorescent TALEs in Arabidopsis thaliana" by Satoru Fujimoto, Shigeo S. Sugano, Keiko Kuwata, Keishi Osakabe and Sachihiro Matsunaga, is published online in the Journal of Experimental Botany.
リンク:
- プレスリリース
- 日経産業新聞「生きた植物 DNA観察 東京理科大 ゲノム編集で発光」(2016.10.20)
- 化学工業日報「蛍光タンパク ゲノム編集で配置 植物DNA生きたまま観察 東京理科大など 作物育種技術に応用期待」(2016.10.20)
- 日経バイオテク「理科大と京大、名大、徳島大、生きた植物の核内DNAを解析できるTALE-FP法 ゲノム編集ツールで植物のエピゲノム育種にも貢献」(2016.10.27)
- 科学新聞「生きたまま植物DNA解析 TALE-FP法 東京理科大グループが開発」(2016.10.28 1面)

関連記事・報道:
2016-10-11

