研究ハイライト
モデル植物シロイヌナズナのゲノム編集効率を 飛躍的に高める手法を開発 〜 植物科学の進展や作物育種への応用に期待 〜
名古屋大学の東山 哲也 教授(名古屋大学 WPI トランスフォーマティブ生命分子研究所 教授)と筒井 大貴 補佐員(名古屋大学 大学院理学研究科)らの研究グループは、モデル植物シロイヌナズナにおいて、狙った遺伝子を高い効率で破壊するゲノム編集の手法を開発しました。
近年、ゲノム編集注1)と呼ばれる、狙った遺伝子を操作(破壊、書き換え)する技術が注目を集めています。その中でも特に、CRISPR/Cas9注2)(クリスパー・キャスナイン)は手順が簡単で生物種を問わず適用が可能なため、ここ数年で急激に利用が広まっています。しかし、植物の中でも特にシロイヌナズナでは編集効率が低く、狙い通りにゲノム編集が行われた個体を作出するためには、多数の個体を用いて選抜しなければならず、時間と手間が必要でした。
研究グループは、RPS5A遺伝子注3)のプロモーター注4)を用いて、Cas9タンパク質を個体発生ごく初期の卵細胞の段階から連続して働かせることで、高効率にゲノム編集を行い、遺伝子を破壊する分子ツールpKAMA-ITACHI Red (pKIR)注5)を開発しました。植物においてタンパク質を作るために従来用いられてきた35Sプロモーター注6)に比べて、飛躍的にゲノム編集の効率が上昇しています。また、pKIRでは、CRISPR/Cas9のための遺伝子を導入した個体を簡便に区別できるよう、その個体の種子に赤色蛍光タンパク質を発現させ、形質転換体の選別効率を上げています。
今後、この手法で狙った遺伝子を破壊することによって、植物の遺伝子の機能の解明に役立つことが期待されます。また、セイヨウアブラナ注7)など、卵細胞に外来遺伝子を導入する形質転換法が利用できる作物に応用し、ゲノム編集の効率化によって育種を加速することも期待されます。
本研究成果は、科学誌「Plant and Cell Physiology」のオンライン速報版で公開されました。
なお、本研究成果は、科学技術振興機構(JST)戦略的創造研究推進事業ERATO「東山ライブホロニクスプロジェクト」(東山 哲也 研究総括)の一環で得られました。
【ポイント】
・ 遺伝子の機能を調べるため、狙った遺伝子を破壊するゲノム編集(CRISPR/Cas9)が用いられてきたが、モデル植物のシロイヌナズナでは効率が低かった。
・ 卵細胞からゲノム編集を行うことで、狙った遺伝子を高い効率で破壊する手法を開発した。
・ 未知の遺伝子群の機能解明が短期間で進展することが期待できる。
・ セイヨウアブラナなどの作物での育種が加速することが期待できる。
研究の内容:
【背景】
ゲノム編集と呼ばれる遺伝子操作技術が注目を集めています。その中でもCRISPR/Cas9と呼ばれる技術は、狙った遺伝子を簡単に破壊もしくは書き換えすることができるために近年、急激に普及しています。この技術は、狙った遺伝子の破壊(ノックアウト)株を作出し、その遺伝子の機能を調べるために植物でも利用されています。しかし、モデル植物であるシロイヌナズナでは、Cas9タンパク質を細胞に発現させる時期の最適化が十分ではなく、個体発生が進んでからCas9タンパク質が働くために組織ごとでノックアウトの度合いが異なるのが問題でした。
実際、植物研究で広く用いられている35Sプロモーターを用いてCas9タンパク質を発現させると、葉ではノックアウトが高頻度で見られるものの、花では検出されづらくなりました。この結果は、標的遺伝子のノックアウト変異率が花の中の生殖細胞で低いことを示しており、変異が次の世代へ遺伝しづらくなるため、ノックアウト変異を持つ第2世代の個体の作出効率が低いことを表しています。
【研究の内容】
上記の問題点を解決すべく、東山教授と筒井補佐員らの研究グループは、Cas9タンパク質を卵細胞の時点からその後の発生過程まで連続して発現させることによって、ゲノム編集を用いた遺伝子のノックアウトを効率化することを目標とし、実験を行いました。
植物が発生、成長して個体を形作る上で最初の細胞が卵細胞、そしてそれが受精して作られるのが受精卵です。卵細胞もしくは受精卵でゲノム編集が行われると、ゲノム編集によって生じた変異が個体全体に受け継がれます。その結果として、次の世代にもその変異が受け継がれ、変異が固定されると考えられます。
本研究では、卵細胞の時点からその後の発生過程まで連続してCas9タンパク質を発現させるためのプロモーターとして、卵細胞の時点から強い活性を維持することが知られるRPS5A遺伝子のプロモーター(RPS5Aプロモーター)を用いました。
まず、PDS3遺伝子のノックアウトを試みました。PDS3遺伝子がノックアウトされると、クロロフィル(葉緑素)が合成されず、植物細胞がアルビノ注8)になることが知られています。RPS5AプロモーターでCas9タンパク質を発現させ、PDS3遺伝子のノックアウトを試みると、第1世代目ですべての葉がアルビノになる個体が得られました(図1)。

図1 PDS3遺伝子のノックアウトによってアルビノになったシロイヌナズナ
RPS5AプロモーターでCas9タンパク質を発現させることにより、効果的にゲノム編集が起こったために、全身でPDS3遺伝子がノックアウトされ、アルビノ化しています。スケールバーは2 mmです。
2番目に出てくる本葉が完全にアルビノである個体の割合は、35SプロモーターでCas9タンパク質を発現させると3.1%だったのに対して、RPS5Aプロモーターでは66.7%であり、多くの個体でPDS3遺伝子がノックアウトされていました。
また、花茎で合成されているクロロフィルa量を調べたところ、従来用いられてきた35SプロモーターでCas9タンパク質を発現させる方法では、花茎生重量1 mgあたりのクロロフィルa量の中央値は650 ngであり、野生株の730 ngとほとんど変わりませんでしたが、RPS5Aプロモーターでは、クロロフィルa量が6.4 ngと大きく低下していました(図2)。このことから、RPS5AプロモーターでCas9タンパク質を発現させることで、ゲノム編集を効率化できると分かりました。
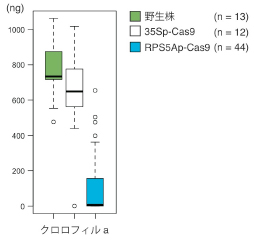
図2 PDS3遺伝子をノックアウトしたシロイヌナズナの花茎におけるクロロフィルaの量
花茎生重量1 mgにおけるクロロフィルaの量を調べました。35SプロモーターでCas9タンパク質を発現させたとき(35Sp-Cas9)は野生株とクロロフィルaの量があまり変わらないのに対して、RPS5AプロモーターでCas9タンパク質を発現させたとき(RPS5Ap-Cas9)は、著しく低下していました。nは調べた植物体の数を表しています。
本研究では、他にAGAMOUS, DUO1, ADH1といった遺伝子のノックアウトも試み、いずれの場合もRPS5AプロモーターによるCas9タンパク質の発現で、遺伝子を効率よくノックアウトできることを示しました。例えば、花の形成に関与するAGAMOUS遺伝子のノックアウト実験では、第1世代において、個体全体でAGAMOUS遺伝子のノックアウトが起こったため、八重咲きの表現型が観察できました(図3)。これは育った12個体のシロイヌナズナすべてで観察されており、非常に高い割合で八重咲きの形質を与えることに成功したといえます。

図3 AGAMOUS遺伝子がノックアウトされたシロイヌナズナ
(A)個体全体で八重咲きの表現型が観察されました。(B)花の拡大写真です。スケールバーは(A)5 cm、(B)1 cmです。
また、ADH1遺伝子のノックアウト実験で、第2世代でノックアウトになっている割合を24系統について調べたところ、その中央値は81%でした(図4)。ノックアウト変異の割合が100%の系統も5系統あったことから、それらの系統では第1世代の生殖細胞(卵細胞と精細胞)すべてでADH1遺伝子がノックアウトされたと考えられます。
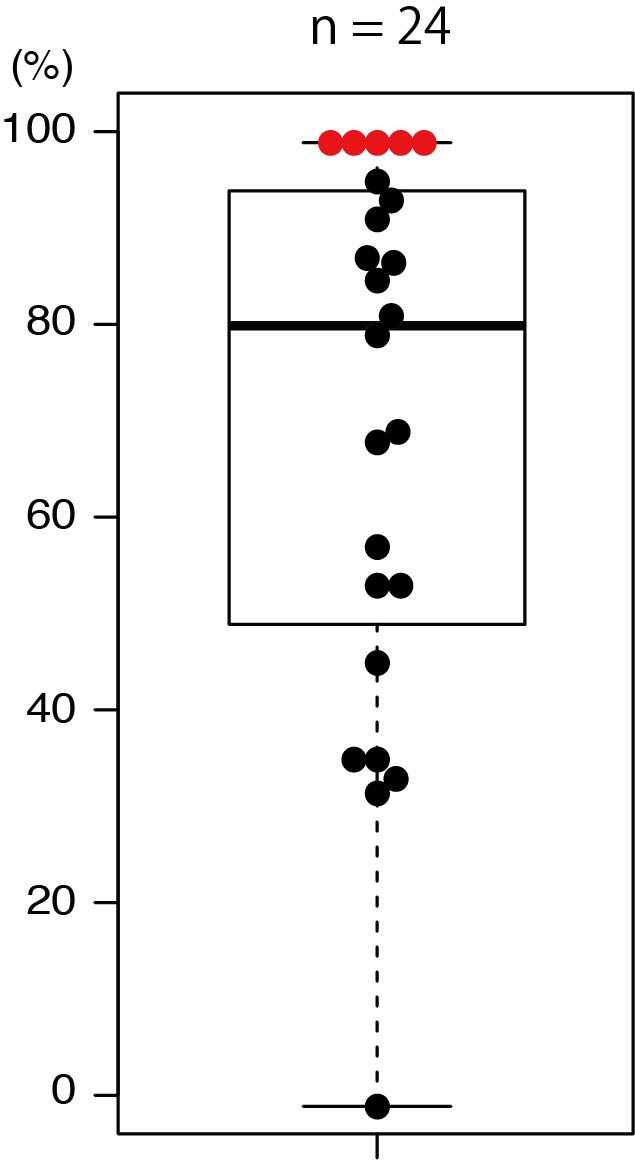
図4 ADH1遺伝子がノックアウトされたシロイヌナズナの割合
24の系統において、それらの第2世代の個体でADH1遺伝子がノックアウトされている割合を調べました。その中央値は81%であり、100%を示す系統も5つ(赤色)得ることができました。
また、第2世代の個体すべてで同じ変異パターンのみが検出された系統もあったことから、この系統では第1世代の受精卵という、発生のごく初期の段階でゲノム編集が起こっていたと考えられます。
また、CRISPR/Cas9のための遺伝子を導入した個体を簡便に区別できるよう、その個体の種子に赤色蛍光タンパク質を発現させています。
以上の結果から、RPS5AプロモーターでCas9タンパク質を発現させることが、効率よくゲノム編集を働かせるのに非常に適した実験系であることが分かりました。現在、この分子ツールをpKAMA-ITACHI Red (pKIR)と名付け(図5)、特許出願中です(特願2016-171590)。
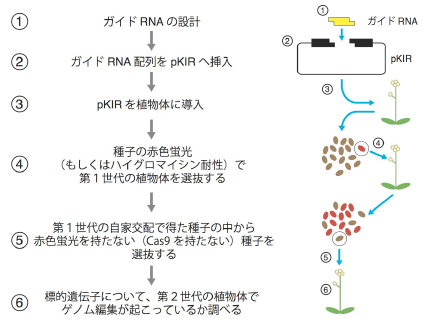
図5 pKIRを用いた実験の手順
pKIRを用いた遺伝子ノックアウト株作出の手順を示しました。Cas9を持つ植物の種子が赤色蛍光タンパク質で標識されているため、Cas9の有無の判断が容易になっています。この手順で進めると、標的遺伝子にノックアウト変異を持っていて、かつCas9を持っていない植物体が第2世代の時点で得られます。実験開始から遺伝子ノックアウト株作出までの期間はおよそ3ヶ月程度となります。
【成果の意義】
従来は遺伝子ノックアウト株の作出に時間と労力がかかっていましたが、本成果を用いると高効率に遺伝子ノックアウト株を作出できるため、研究の時間短縮、低コスト化が図れると期待できます。
特に、機能が重複している遺伝子群を解析する場合、それらの既存の変異体同士を交配することで多重遺伝子ノックアウト株を作る必要がありましたが、pKIRを用いれば容易に作出できるようになります。これによって、これまで未知であった遺伝子群の機能解明が期待できます。
また、シロイヌナズナと同様に、セイヨウアブラナなどのアブラナ科植物では、花序浸し法注9)によって卵細胞に遺伝子を導入して形質転換を行う手法が報告されています。pKIRをこれらの植物に利用することで高効率なゲノム編集が可能であると推測でき、育種の加速が期待できます。
【用語説明】
注1)ゲノム編集
DNA切断酵素を用いて、染色体上の任意のDNA配列を自在に操作(書き換え、破壊など)することのできる技術。これまで開発されてきたZFN(ジンクフィンガーヌクレアーゼ)、TALEN(タレン)に代わって、近年はより簡便なCRISPR/Cas9の利用が広まりつつあります。
注2)CRISPR/Cas9
Clustered Regularly Interspaced Short Palindromic Repeats/CRISPR associated protein9の略で、DNA切断活性を持つCas9タンパク質と、1本鎖RNAであるガイドRNAという2つの要素だけで、染色体上の任意の場所をゲノム編集することができる新規の遺伝子改変技術。
注3)RPS5A遺伝子
Ribosomal Protein Subunit 5Aの略。リボソームタンパク質のひとつをコードしている遺伝子。
注4)プロモーター
遺伝子がいつ、どの細胞で発現するかを決めているDNA配列。この配列の下流に任意の遺伝子配列をつなぐことで、特定の時期に特定の細胞で遺伝子を発現させることができます。
注5)pKAMA-ITACHI Red (pKIR)
種子で赤色蛍光タンパク質を発現させるためのDNA配列を含む環状DNA(プラスミドDNA)であるpFAST-R01(甲南大学 西村いくこ教授らによって開発)に、RPS5AプロモーターでCas9タンパク質を発現させるためのDNA配列と、ガイドRNAを発現させるためのDNA配列を挿入したものです。
注6)35Sプロモーター
植物において、遺伝子を過剰発現させるときによく使われるプロモーターのひとつ。胚発生後期から遺伝子を発現させることができますが、卵細胞や受精卵では活性が検出できないことが知られています。
注7)セイヨウアブラナ
学名 Brassica napus。アブラナ科の植物で、菜種油の原料として広く栽培されています。
注8)アルビノ
クロロフィル(葉緑素)などの色素を持たないために白く見える現象の呼称。
注9)花序浸し法
主にシロイヌナズナで広く用いられている形質転換方法。導入したい遺伝子を持つアグロバクテリウムの溶液にシロイヌナズナのつぼみを浸し感染させることで、卵細胞に遺伝子を導入することができます。
論文情報:
This article "pKAMA-ITACHI vectors for highly efficient CRISPR/Cas9-mediated gene knockout in Arabidopsis thaliana" by Hiroki Tsutsui and Tetsuya Higashiyama is published online in Plant and Cell Physiology.
リンク:
- プレスリリース
- JST「モデル植物シロイヌナズナのゲノム編集効率を 飛躍的に高める手法を開発」(2016.11.18)
- 日本の研究.com「モデル植物シロイヌナズナのゲノム編集効率を 飛躍的に高める手法を開発」(2016.11.18)
- AlphaGalileo JP 「モデル植物シロイヌナズナのゲノム編集効率を 飛躍的に高める手法を開発」(2016.12.05)
- EurekAlert! JP 「モデル植物シロイヌナズナのゲノム編集効率を 飛躍的に高める手法を開発」(2016.12.05)
- 日経産業新聞「植物の遺伝子 効率改変 名大、ゲノム編集の新手法」(2016.11.22 朝刊 8面)



東山 哲也 教授 筒井 大貴 補佐員
関連記事・報道:
2016-12-05

